SARS-CoV-2: una nueva amenaza
-
Cristina A. López Rodríguez
Resumen
Objetivos
Aportar una breve revisión del conocimiento actual sobre el virus SARS-CoV-2, cuya irrupción ha causado un gran impacto en la sociedad a escala mundial.
Contenido
Esta revisión aporta una visión general de diversos aspectos del virus y de la respuesta inmunitaria que desencadena, así como aproximaciones diagnósticas y terapéuticas que se están llevando a cabo.
Resumen
SARS-CoV-2 es un virus RNA con características peculiares que lo diferencian de sus predecesores SARS-CoV y MERS. Dada sus características estructurales y su patogenia, es capaz de provocar diversas manifestaciones clínicas según avanza la enfermedad. Se ha demostrado que el sistema inmunitario juega un papel importante en la respuesta frente a este virus y, por ende, es crucial el estudio de los anticuerpos y las poblaciones linfocitarias durante los distintos estadios de la enfermedad.
Perspectiva
El conocimiento del efecto del virus y la respuesta inmunitaria es determinante para el desarrollo de vacunas, terapias y técnicas diagnósticas de calidad, esenciales para el control y la erradicación de la enfermedad.
Introducción
SARS-CoV-2
A finales del mes de Diciembre del 2019, se reportaron nuevos casos de neumonía de causa desconocida cuyo foco se encontraba en un mercado mayorista de mariscos y animales en Wuhan, provincia de Hubei, China [1].Tras el repunte de estos casos, se empezó a analizar muestras del líquido broncoalveolar (BAL) de pacientes con neumonía y síntomas compatibles, con el fin de estudiar el material genético y obtener la secuenciación genómica del agente infeccioso. Finalmente se detectó un nuevo coronavirus, SARS-CoV-2 siendo identificado como β-coronavirus, con características similares pero distinto de otros virus causantes de epidemias como el SARS y el MERS [1].
Características del nuevo SARS-CoV-2
Los Coronavirus (CoV) pertenecen a la familia Coronaviridae, los cuales son virus RNA envueltos, monocatenarios de 26 a 32 kilobases de longitud [2].
Estos virus pueden causar una amplia gama de enfermedades tanto respiratorias como entéricas, hepáticas y neurológicas. Los coronavirus se dividen en cuatro subfamilias: α, β, γ y δ-CoV. Estas infecciones humanas por CoV son causadas por α y β-CoV. Otros β-CoV, como son el SARS-CoV y MERS-CoV, conducen a infecciones respiratorias graves y potencialmente mortales [3].
La estructura del SARS-CoV-2 es similar a los otros β-CoVs: El virión presenta una nucleocápside constituida por ARN y la proteína fosforilada de la nucleocápside (N). Esta nucleocápside se encuentra recubierta de una doble capa de fosfolípidos donde se encuentran dos proteínas “espiga”: la glicoproteína trimérica de espiga (S) y la hemaglutinina esterasa (HE) [3]. Ésta última sirve como una enzima destructora de receptores que facilitará la liberación del virión de las células infectadas y permitirá escapar de la unión a las células huésped no permisivas [4]. La proteína S es una proteína de fusión viral que promueve la unión de las membranas viral y celular durante la entrada. Ésta también suele ser el antígeno principal de los anticuerpos neutralizantes generados durante la infección y uno de los focos del diseño de vacunas [4].
A lo largo de la cubierta, también se encuentran las proteínas de membrana (M) y envuelta (E) [3] (Figura 1).
![Figura 1:
Estructura del SARS-CoV-2. Adaptado de Jin et al. [3].](/document/doi/10.1515/almed-2020-0045/asset/graphic/j_almed-2020-0045_fig_001.jpg)
Estructura del SARS-CoV-2. Adaptado de Jin et al. [3].
Patogénesis
El SARS-CoV-2 utiliza el receptor ACE2 (enzima convertidora de angiotensina 2) para su entrada en el huésped, al igual que SARS-CoV [5]. Este tipo de receptor se encuentra localizado en los pulmones, corazón, riñones e intestino [3] y mayoritariamente asociado a enfermedades cardiovasculares como la hipertensión, donde se ha observado una mayor expresión de éstos por mecanismo compensatorio de la propia enfermedad [6].
El virión se une al receptor ACE2 mediante la glicoproteína S. La glicoproteína S incluye dos subunidades, S1 y S2. La subunidad S1 determina el tropismo celular mediante el dominio de unión al receptor (RBD) mientras que la S2 media la fusión de la membrana celular del virus gracias a dos dominios en tándem (HR1 y HR2) [7].
Después de la unión al receptor, se produce un cambio conformacional en la proteína S, facilitando la fusión de la envoltura viral con la membrana celular a través de la vía endosómica. A continuación, el SARS-CoV-2 libera ARN en la célula huésped. El ARN del genoma se traduce en poliproteínas de replicasa viral pp1a y 1ab, que a su vez se dividen en pequeños productos mediante proteinasas virales. La polimerasa produce una serie de ARNm por transcripción discontinua y finalmente se traduce en proteínas virales. Las proteínas virales y el ARN del genoma se ensamblan en viriones en el retículo endoplásmico y en el aparato de Golgi, se transportan a través de vesículas y se liberan de la célula [8].
Etapas de la infección y características clínicas:
En varios estudios como el de Siddiqi HK et al., se proponen tres estadios clínicos de la enfermedad [9]
Etapa I: Establecimiento temprano de la enfermedad.
Esto implica un período de incubación de entre 1 a 14 días, principalmente de 3 a 7 días, asociado con síntomas leves y a menudo no específicos, como malestar general, fiebre y tos seca [7]. En esta etapa, el SARS-CoV-2 se multiplica y establece su residencia en el huésped, centrándose principalmente en el sistema respiratorio. Durante la etapa de incubación el paciente es contagioso y el virus puede transmitirse a través de gotículas, secreciones respiratorias y contacto directo [7]. Las alteraciones de laboratorio más frecuentes en esta fase son la elevación de la proteína C reactiva (PCR), enzimas hepáticos y ligeras linfopenia y neutrofilia [9].
Etapa II (moderada): compromiso pulmonar (IIa) sin y (IIb) con hipoxia.
Durante la segunda semana, hasta un 80% de pacientes pueden desarrollar una neumonía viral, con tos, disnea, fiebre y posiblemente hipoxia (definido como un PaO2 / FiO2 de <300 mmHg). A nivel radiológico, se observan infiltrados bilaterales u opacidades en vidrio esmerilado tanto en las imágenes con radiografía de tórax como en tomografía axial computarizada. Los análisis de sangre revelan linfopenia, junto con transaminitis. Los marcadores de inflamación sistémica (IL-6, velocidad de sedimentación globular (VSG), PCR, dímero D y ferritina) se encuentran elevados, pero no excesivamente [9].
Etapa III (severa): Hiperinflamación sistémica.
Esta es la etapa más grave de la enfermedad a la que llega aproximadamente un 15% de pacientes y que se manifiesta como un síndrome de hiperinflamación sistémica extrapulmonar. En esta etapa, los marcadores de inflamación sistémica (entre ellos la IL-6) están muy elevados e incluso se puede llegar a desarrollar una forma similar a la linfohistiocitosis hemofagocítica (SHLH). Los pacientes con enfermedad más grave también presentan una elevación de citocinas proinflamatorias, dímero D, ferritina, troponinas y prohormona N-terminal del péptido natriurético cerebral (NT-proBNP) además de una marcada neutrofilia y linfopenia. También puede desarrollar shock, vasoplejía, insuficiencia respiratoria e incluso colapso cardiopulmonar. Por último, puede llegar a dar un fallo multiorgánico [9].
Respuesta inmunitaria
Respuesta celular
La respuesta inmunitaria en los pacientes con COVID-19 se puede dividir en tres fases, las cuales se solapan con las etapas clínicas anteriormente descritas:
Fase de viremia. En esta fase la presencia del ARN viral en sangre aumenta exponencialmente durante la primera semana. La respuesta inicial de tipo innato, con producción de interferones tipo-I (IFN)-I en las vías aéreas es clave para el control de la replicación viral y la puesta en marcha de una correcta respuesta celular inmunitaria efectiva [10].
Fase aguda o de neumonía. Tiene lugar a los 7-10 días del inicio de la infección. Se caracteriza por un estado inflamatorio con neutrofilia, aumento de citocinas proinflamatorias y quimiocinas (interleucina (IL)-6, factor de necrosis tumoral (TNF)-α, IFN-I y -II, IL-8, CXCL-10), disminución de linfocitos T y B en sangre periférica y alteración de diversos parámetros bioquímicos y hematológicos, cómo incremento de dímero D, ferritina, lactato deshidrogenasa (LDH), PCR y disminución de la albúmina 56 [11]. Durante esta etapa la carga viral ha alcanzado su nivel máximo y comienza a disminuir. Llegados a este punto, el paciente puede evolucionar de manera diferente en función de la capacidad de su sistema inmunitario de controlar la infección viral.
Fase de recuperación. Si la respuesta inmunitaria del paciente es suficiente para hacer frente a la infección y la carga viral disminuye paulatinamente, los marcadores de inflamación se reducen y las poblaciones linfocitarias se recuperan [12].
Fase severa. En caso de que el sistema inmunitario no sea capaz de controlar la infección, el número de linfocitos disminuye aún más mientras que la carga viral y los marcadores de inflamación siguen aumentado. Los pacientes en este estadio empeoran progresivamente pudiendo llegar a morir [12].
Aunque los mecanismos subyacentes a este comportamiento inflamatorio del sistema inmunitario son poco conocidos, se han identificado algunos procesos celulares que nos pueden ayudar a comprender la evolución de la enfermedad.
Por lo que se refiere a la respuesta inicial frente al virus, una expresión de IFN-I poco potente o retrasada en el tiempo se ha asociado a un peor pronóstico de la enfermedad [10]. En este sentido, se ha descrito que el propio virus es capaz de disminuir la expresión de genes de IFN-α e IFN-β en monocitos durante la infección [13]. Esto implica una reducción de la respuesta antiviral asociada a estas moléculas y de la expresión de otras moléculas como las del complejo mayor de histocompatibilidad (MHC) de clase I, catepsinas, proteínas lisosomales y del proteasoma, todas ellas implicadas en la presentación de antígenos y la respuesta inmunitaria de tipo 1 (antiviral) [13], [14].
Algunos pacientes COVID-19 presentan una liberación masiva de citocinas llamada “Tormenta de citocinas”, proceso que se asemeja al que ocurre en otras patologías como el SHLH. Las causas exactas que inducen esta liberación no se conocen, aunque se postula que el bloqueo primario de la respuesta de IFN-I puede producir de alguna manera una respuesta secundaria exagerada de otras citocinas [15]. Las citocinas que se pueden observar elevadas son la IL-6, el TNF-α, el IFN-γ o la IL-8, entre otras [15], [16]. La IL-1β, una de las principales citocinas pro-inflamatorias, se encuentra en niveles normales en la sangre periférica de estos pacientes, ya que ésta tiene una semivida corta y suele degradarse rápidamente. Este aspecto se tiene que tener en cuenta a la hora de monitorizar los pacientes con tratamientos biológicos, dado que la IL-1β no es un buen parámetro para monitorizar [17].
Por lo que respecta a las subpoblaciones linfocitarias, en los pocos estudios realizados hasta el momento los linfocitos T CD4+ y CD8+ son las poblaciones que presentan una mayor disminución en cuanto a número, seguidas por los linfocitos NK y B. Este descenso parece acentuarse en los pacientes más graves y en aquellos con mayores niveles de citocinas proinflamatorias. También se ha observado una disminución en los linfocitos T reguladores [18].
Una vez llegados a la fase aguda, los pacientes que empiezan a controlar el virus son aquellos que consiguen desarrollar una respuesta inmunitaria específica adecuada, tanto humoral como celular [19]. En este sentido, se han observado incrementos en sangre periférica de los porcentajes de plasmablastos, productores de anticuerpos, (CD19+CD27highCD38high) y linfocitos T colaboradores foliculares (T folicular helper -Tfh-) circulantes (CD4+CXCR5+ICOS+PD-1+) a partir del día 7 de desarrollar la clínica [20]. También se ha visto un incremento de linfocitos T CD8+ con fenotipo activado (CD38+HLA-DR+) en pacientes con una buena evolución y en aquellos que responden a los tratamientos inmunomoduladores [21]. Este aumento no se aprecia en los pacientes más graves, los cuales además presentan mayor cantidad de linfocitos T CD8+ con fenotipo exhausto (PD-1+ CTLA-4+ TIGIT+) [22].
La respuesta celular que se desarrolla es fundamentalmente de tipo 1. Estudios en pacientes infectados por el SARS-CoV han demostrado la presencia de linfocitos T CD4+ y CD8+ productores de IFN-γ específicos frente a diversos antígenos virales [23]. Se han descrito poblaciones T CD4+ de memoria frente al SARS-CoV residentes en las vías aéreas incluso 11 años después de la infección [24]. Estas poblaciones celulares son capaces de reaccionar frente al virus rápidamente, liberando IFN-γ, promoviendo el desplazamiento de células dendríticas pulmonares cargadas con el antígeno a los nodos linfáticos mediastínicos y atrayendo linfocitos T CD8+ específicos frente al virus al pulmón [23], [24], [25]. Hacen falta más estudios en sangre periférica de subpoblaciones linfocitarias minoritarias que permitan monitorizar estos cambios durante la evolución de la enfermedad.
Respuesta humoral
Al tratarse de una enfermedad producida por un nuevo virus, existe poco conocimiento acerca de la respuesta humoral que se produce en los pacientes infectados. En este contexto, es importante conocer tanto la cinética de seroconversión, los títulos de los anticuerpos, la duración en el plasma de los pacientes una vez superada la infección y su capacidad como anticuerpos neutralizantes.
Por ello, algunos autores se han centrado en la respuesta humoral de otros virus con los que el SARS-CoV-2 presenta cierta similitud, como el SARS-CoV. En el estudio realizado por Li et al. [26], analizaron la respuesta inmune humoral de 20 pacientes en donde pudieron observar la cinética de aparición de las inmunoglobulinas desde la semana de inicio de la enfermedad hasta la semana 12 [26]. Destacaron la positividad de anticuerpos IgG a partir de la semana 3, pudiéndose mantener hasta 3 meses tras los primeros síntomas. Estos anticuerpos reconocían mayoritariamente a las proteínas S y N del virus [27]. Por otro lado, los anticuerpos tipo IgM aparecían en los primeros días y desaparecían progresivamente hasta la semana 12. Ante estos resultados, los autores sugieren que los anticuerpos IgG contra el SARS-CoV pueden jugar un papel importante en la protección frente a la infección por SARS [27].
Más recientemente estudios con el nuevo SARS-CoV-2 han podido demostrar una cinética similar, a pesar de que no en todos ellos se analizan los mismos antígenos o el mismo tipo de pacientes.
Guo et al. [28] detectaron la aparición de anticuerpos IgM e IgA a los 5 días del inicio de los síntomas, pudiéndose detectar hasta 3 semanas más tarde. Por otro lado, la presencia de IgG se detectó alrededor de los 14 días después del inicio de la clínica, aunque algunos autores apuntan a que la IgG podría detectarse antes, alcanzando su plateau a los 21 días, existiendo variación según paciente [29], [28]. [Figura 2]
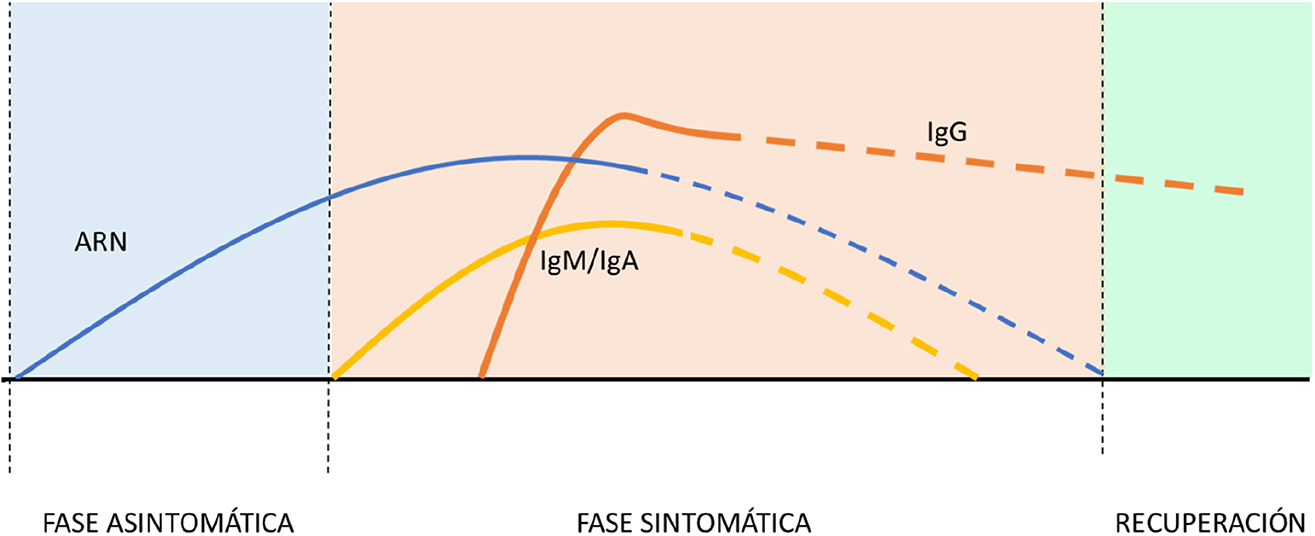
Representación de la cinética de ARN y anticuerpos IgG, IgA e IgM anti SARS-CoV-2 en las diferentes fases clínicas de la enfermedad COVID19.
Estos resultados son similares a los reportados por otros autores como Theravajan et al. que analizaron la cinética de la respuesta inmunológica en un paciente australiano con SARS-CoV-2 [20].
Estudios como el de Zhao et al. [30], observaron una cinética de aparición de anticuerpos totales, IgM e IgG a 11, 12 y 14 días respectivamente, alcanzando una seropositividad del 50% en el día 11 y un 100% a día 39 [30].
La aparición de títulos elevados de IgG antes de los 14 días se ha relacionado con la severidad de la enfermedad [30], [31]. La causa exacta de este fenómeno se desconoce actualmente, aunque se cree que niveles mayores de anticuerpos conducen a un incremento de la formación de inmunocomplejos fijadores del complemento en los tejidos pulmonares, con la consiguiente inflamación y daño tisular [32]. También se sospecha de la existencia de anticuerpos relacionados con el fenómeno ADE (antibody-dependent enhancement), los cuales se han observado en el SARS-CoV. Este fenómeno se caracteriza por la unión de los anticuerpos IgG al virus [33], [34]. El inmunocomplejo virus-IgG se uniría por la fracción constante (Fc) de las IgG a los receptores Fc gamma receptor (FcγR) presentes en la superficie de monocitos, macrófagos y linfocitos B. La unión Fc- FcγR facilitaría la entrada del virus en estas células. Esta infección de las células no daría lugar a una replicación viral funcional pero induciría la activación de las células infectadas y la generación de citocinas pro-inflamatorias como el TNF-α y la IL-6 incrementando la inflamación de los pacientes [34].
Técnicas de diagnóstico en el laboratorio
Con la nueva aparición de casos diagnosticados de COVID-19, empezaron a desarrollarse técnicas diagnósticas en el laboratorio para su detección precoz y posible relación con el estadio de inmunización de los pacientes infectados.
Actualmente, se disponen de diversas técnicas para el diagnóstico de laboratorio del COVID-19:
Técnicas de detección de ácidos nucleicos: Esta técnica se basa en la llamada RT-PCR, o reacción en cadena de la polimerasa con transcriptasa inversa, que consiste en la amplificación del genoma del virus y es actualmente la técnica Gold Standard. Para esta técnica, se recomienda recoger muestras tanto del tracto respiratorio superior como inferior para una mayor sensibilidad en la prueba [35]. La ventaja es la alta especificidad y sensibilidad que presenta (sobre todo dentro de los 7 primeros días del inicio de los síntomas) y en el procesamiento de gran número de muestras a la vez. Es cierto que en algunos casos puede dar falsos negativos [36], [37] debido a errores preanalíticos (recogida inadecuada de la muestra, mala conservación) [38] o por baja carga viral en la muestra a testar [39]. La determinación de la presencia del virus por RT-PCR puede perder sensibilidad a lo largo del tiempo, llegando a alcanzar una sensibilidad menor al 50% después del día 14 de inicio de los síntomas. Por ello, se han propuesto técnicas complementarias como la determinación de los anticuerpos para clasificar a estos pacientes [28].
Técnicas de detección de antígeno: Se basa en la detección de antígenos propios del virus (normalmente la proteína S o la N) pero no existen muchos estudios acerca de la sensibilidad y especificidad que ofrecen este tipo de pruebas. En el estudio llevado a cabo por Diao et al., [40] mostraron que en muestras de pacientes que habían sido positivas por RT-PCR con Ct (Ciclo umbral) <40, la sensibilidad era del 68% y la especificidad del 100%, mientras que con Ct < 30, la sensibilidad era del 98% y especificidad del 100% [40]. La principal ventaja es el tiempo de procesamiento, ya que se trata de ensayos de inmunocromatografía (test rápidos o llamados Point Of Care) cuyo resultado se obtiene en unos 15-30 minutos. No obstante, su utilidad en la práctica diaria es aún controvertida.
Técnicas de detección de anticuerpos: Consiste en la detección de anticuerpos tipo IgA, IgM o IgG en pacientes infectados por COVID-19. Existen técnicas semicuantitativas (ensayo tipo ELISA o CLIA) o cualitativas (inmunocromatografía, Point of Care) para su detección. Dado que la respuesta inmune es detectable principalmente entre 7 y 11 días después de la exposición al virus aunque algunos pacientes pueden desarrollar anticuerpos antes [38], esta prueba no debe realizarse la primera semana desde el inicio de síntomas por baja sensibilidad. Sin embargo, a partir del día 7, la determinación conjunta de RT-PCR y anticuerpos puede incrementar la sensibilidad diagnóstica hasta un 95% [28]. A partir del día 7 de inicio de los síntomas, la sensibilidad y especificidad que pueden alcanzar estas pruebas mediante inmunocromatografía, pueden llegar a ser de un 88% y 90,6% respectivamente [41]. Los resultados de los estudios realizados mediante ELISA, fueron muy similares [42]. No obstante, al igual que con las técnicas de detección de antígeno, son necesarios más estudios para determinar la sensibilidad y especificidad de las diferentes técnicas [42].
Por otro lado, se ha observado que la combinación de RT-PCR y detección de anticuerpos específicos incrementa el porcentaje de detección hasta el 100% a partir de los 15 días del inicio de los síntomas [28], [29].
Las ventajas y desventajas de las técnicas disponibles en el laboratorio se resumen en la Tabla 1.
Ventajas y desventajas de las técnicas disponibles en el laboratorio para el diagnóstico de infección por SARS-CoV-2.
| Ventajas | Desventajas | |
|---|---|---|
| RT-PCR |
|
|
| Antigeno viral |
|
|
| Anticuerpos |
|
|
Importancia de la detección de anticuerpos
La determinación de anticuerpos puede ser de utilidad para la confirmación del diagnóstico de la infección, de manera complementaria a otras herramientas como la RT-PCR , disminuyendo el porcentaje de falsos negativos y, consecuentemente, aumentando la sensibilidad diagnóstica [28]. Además ayudan a evaluar el estadio en el que se encuentra el paciente (Tabla 2) [44].
Interpretación de resultados de RT-PCR junto a anticuerpos IgA/IgM e IgG en infección por SARS-CoV-2. Adaptado de [43].
| Resultado | Significado clínico | ||
|---|---|---|---|
| PCR | IgA/IgM | IgG | |
| − | − | − | Negativo |
| + | − | − | Periodo ventana |
| + | + | − | Fase temprana de la infección |
| + | + | + | Fase activa de la infección |
| + | − | + | Fase final de la infección |
| − | + | − | Fase temprana con falso negativo. Realizar PCR de confirmación |
| − | + | + | Enfermedad en evolución. Realizar PCR de confirmación |
| − | − | + | Infección pasada y curada |
Por tanto, teniendo en cuenta lo dicho anteriormente y atendiendo al documento propuesto por la Sociedad Española de Inmunología [44], la detección de Ac anti-SARS-CoV-2 también serían de utilidad en pacientes con clínica compatible con COVID-19 y RT-PCR negativa.
No existe mucha información sobre la inmunidad protectora que se puede desarrollar frente al SARS-CoV-2. Actualmente, sólo existe un estudio que analiza esta inmunidad en el mono Rhesus [45]. En este trabajo, se observó que los monos que son capaces de generar un título elevado de anticuerpos neutralizantes en una etapa temprana de la infección, posteriormente no se reinfectan con el virus. Por tanto, aunque hacen falta más estudios, estos resultados apoyan la gran utilidad de la determinación de anticuerpos a nivel de la población general para detectar los individuos que hayan pasado la infección de forma asintomática, así como aquellos que hayan podido generar inmunidad protectora [44].
Reactivos comerciales para la detección de antígenos y anticuerpos
Actualmente, se dispone de una gran variedad de reactivos comerciales, muchos en periodo de validación y otros con certificado CE (Conformidad Europea), para la detección tanto de antígenos como de anticuerpos anti-SARS-CoV-2 por diferentes técnicas. Éstos se han descrito y comparado de manera exhaustiva en el repositorio “Foundation for Innovative New Diagnostics (FIND)” [46].
No obstante, es recomendable realizar validaciones antes de su implantación a nivel rutinario en los laboratorios. Además, debido a la gran demanda durante la pandemia, a nivel práctico, antes de seleccionar un reactivo debe considerarse su disponibilidad y periodo de entrega.
En un estudio reciente, dirigido por Lassaunière et al., se analizaron diferentes test serológicos (dos ELISA y seis inmunocromatografías Point of Care) de distintas empresas con diferentes especificidades y sensibilidades y se realizó una comparativa entre los métodos utilizados y sus validaciones internas, facilitando así la elección del test serológico [47]. Así mismo, en Estados Unidos se ha iniciado el “COVID-19 testing project”, un proyecto para evaluar y comparar las características de los reactivos que existen en el mercado [48]
También se están desarrollando inmunoensayos de quimioluminiscencia (CLIA) en plataformas automatizadas. Existen pocos estudios que utilicen esta técnica como test diagnóstico, pero autores como Lin et al. [49], en una comparativa entre la técnica ELISA y CLIA, observaron una mayor sensibilidad y especificidad en el ensayo de quimioluminiscencia.
Aunque COVID-19 es una nueva enfermedad, es importante la validación previa a su utilización para poder valorar posibles reacciones cruzadas con otros coronavirus y obtener un resultado óptimo en el diagnóstico y seguimiento de la enfermedad.
Tratamiento de COVID-19
Actualmente, apenas dos meses tras el inicio de la pandemia en nuestro país, no existe una aproximación terapéutica establecida. Existe gran número de ensayos clínicos que intentan identificar/reposicionar fármacos para tratar el COVID-19, por lo que el abanico terapéutico cambia continuamente.
Los fármacos utilizados en el tratamiento de los pacientes COVID-19 se pueden dividir en dos grandes grupos: los que intentan frenar la replicación viral y aquellos que tienen como objetivo reducir la hiper-inflamación sistémica [50].
Entre los fármacos que bloquean la replicación viral más estudiados podemos encontrar:
Lopinavir/ritonavir. El lopinavir es un inhibidor de la proteasa del VIH que ha sido utilizado ampliamente por las autoridades chinas en el tratamiento de pacientes COVID-19 [51].
Remdesivir. Se trata de un análogo nucleotídico que interfiere en la polimerización del ARN viral. Aunque hay estudios que asocian este fármaco a un beneficio clínico aún se requiere de una mayor evidencia científica para su uso generalizado en pacientes COVID-19 [52]
Hidroxicloroquina/cloroquina. Estos fármacos antipalúdicos han demostrado tener cierta capacidad antiviral in vitro frente al SARS-CoV-2. Se ha observado que interfieren tanto en la entrada como en la replicación del virus. También pueden interferir en la señalización de los TLR (toll like receptors), reduciendo así la activación inmunitaria [53]. A pesar de estas características, su beneficio clínico es aún controvertido [54].
En relación con los fármacos utilizados para reducir la hiper-inflamación, en esta enfermedad, hay que tener en cuenta cómo funciona la respuesta inmunitaria inicial a cualquier patógeno. En ella los macrófagos, granulocitos, células dendríticas y las células epiteliales y endoteliales son capaces de detectar la presencia de proteínas y ARN/ADN del patógeno a través de receptores tipo TLR y NLR (NOD like receptors), entre otros receptores. La activación de estos receptores induce la liberación de IL-1β e IL-6. Estas citocinas a su vez inician una cascada de inflamación que implica la producción de otras citocinas y quimiocinas, la inducción de fiebre, la producción de reactantes de fase aguda en el hígado y la generación de leucocitos en la médula ósea [55], [56].
Así, dentro del grupo de fármacos cuyo objetivo es reducir la hiper-inflamación se han utilizado diferentes tratamientos, con distinta especificidad en cuanto a su mecanismo de acción:
Corticoides. Han sido ampliamente utilizados en pacientes con COVID-19. Sin embargo, su utilidad no ha sido claramente demostrada. Existen varios estudios que indican que estos fármacos no sólo no son beneficiosos, sino que incluso pueden ser perjudiciales, aunque existen datos controvertidos [54], [57]. Actualmente la Agencia Española del Medicamento y Productos Sanitarios (AEMPS) desaconseja su uso y lo restringe a situaciones puntuales [57], [58].
Inhibidores de JAK [Jakinibis]. Los inhibidores de JAK [Janus Kinase] son fármacos que bloquean la transmisión de la señal de activación de los receptores de numerosas citocinas (IL-6, IL-4, IL-17, IL-12, IL-10, etc.) y la consiguiente activación de los factores de transcripción STAT. Con ello se consigue una inhibición del efecto de la cascada de citocinas a diferentes niveles [59].
Anticuerpos monoclonales: Atendiendo al papel clave de las citocinas IL-1 y IL-6 en la cascada de inflamación, se han ensayado algunos anticuerpos que tratan de bloquear su efecto en los pacientes COVID-19:
Tocilizumab y sarilumab. El tocilizumab es un anticuerpo anti- receptor de la IL-6, que impide la unión de la IL-6 a su receptor, bloqueando así su acción sobre la célula diana. El sarilumab es un anticuerpo inhibidor de la IL-6. La AEMPS aconseja el uso de estos fármacos siempre que los niveles de IL-6 se encuentren por encima de 40 pg/ml [60], [58].
Anakinra. Se trata de una versión recombinante de un antagonista fisiológico del receptor de la IL-1 [61].
Otras aproximaciones que se barajan para el tratamiento de pacientes con COVID-19 son el uso de plasma hiperinmune de pacientes que hayan pasado la infección [62] o de anticuerpos monoclonales bloqueantes del virus [63]. Todos estos tratamientos se encuentran en fases de estudio y todavía carecen de una evidencia científica sólida [54].
Tal y como se ha comentado previamente, la utilización de diferentes terapias frente al COVID-19 se encuentra en continua revisión, y se publican periódicamente actualizaciones por la AEMPS [64].
Búsqueda de la vacuna: pasado, presente y futuro
Desde la irrupción del SARS-CoV-2 se han puesto en marcha numerosas investigaciones y ensayos clínicos para desarrollar vacunas seguras y eficaces contra el virus [65].
Basándose en la experiencia de otros coronavirus como el SARS y el MERS, se están desarrollando posibles vacunas para SARS-CoV-2, dada su similitud estructural y genómica [66]. Las estrategias utilizadas incluyen el uso proteínas enteras del virus, péptidos virales, ARN mensajero (ARNm), o el propio virus inactivado.
Cabe destacar la necesidad de encontrar el antígeno adecuado capaz de generar una respuesta inmunitaria protectora, segura y duradera. En este sentido, la proteína S (espiga) del virus es la que ha demostrado, en estudios con SARS-CoV, tener una mayor capacidad de desarrollar tanto una respuesta humoral como celular efectivas [66]. La formación de anticuerpos neutralizantes frente a este antígeno implicaría el bloqueo de la unión del virus al receptor ACE2 y la consiguiente infección de la célula diana [67].
Las vacunas basadas en vectores virales ofrecen un alto nivel de expresión de proteínas y estabilidad a largo plazo, e inducen fuertes respuestas inmunes. El uso de adyuvantes también podría mejorar la inmunogenicidad [68] y prolongar la respuesta humoral [69]. También hay que tener en cuenta la generación de una respuesta inmunitaria celular de memoria para el desarrollo de estas vacunas [69]
Actualmente, existen más de 90 proyectos en marcha para la generación de una vacuna, algunos de ellos ya en fases clínicas iniciales [69], [70].En este momento, en el campo de la investigación y el desarrollo de tratamientos y vacunas contra el nuevo SARS-CoV-2 se están realizando grandes esfuerzos a nivel internacional. En los próximos meses podremos observar cómo evolucionan los distintos proyectos y en qué medida estas terapias se podrán aplicar a la población.
-
Nota del artículo: La versión traducida del artículo puede encontrarse aquí: https://doi.org/10.1515/almed-2020-0083
-
Financiación de la investigación: No declarada.
-
Contribución de los autores: Todos los autores han aceptado la responsabilidad del contenido completo del manuscrito ya prueban su envoi.
-
Conflicto de intereses: Los autores declaran no tener ningún conflicto de intereses.
Referencias
1. Zhu, N, Zhang, D, Wang, W, Li, X, Yang, B, Song, J, et al. A novel coronavirus from patients with pneumonia in China, 2019. N Engl J Med 2020;382:727–33. https://doi.org/10.1056/nejmoa2001017.Search in Google Scholar PubMed PubMed Central
2. Zheng, J. SARS-CoV-2: an emerging coronavirus that causes a global threat. Int J Biol Sci 2020;16:1678–85. https://doi.org/10.7150/ijbs.45053.Search in Google Scholar PubMed PubMed Central
3. Jin, Y, Duan, G, Yang, H, Ji, W, Wu, W, Chen, S, et al. Virology, epidemiology, pathogenesis, and control of COVID-19. Viruses 2020;12:1–17. https://doi.org/10.3390/v12040372.Search in Google Scholar PubMed PubMed Central
4. Tortorici, MA, Veesler, D. Structural insights into coronavirus entry. Adv Virus Res 2019;105:93–116. https://doi.org/10.1016/bs.aivir.2019.08.002.Search in Google Scholar PubMed PubMed Central
5. Zhou, P, Yang, XL, Wang, XG, Hu, B, Zhang, B, Zhang, W, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature 2020;579:270–3. https://doi.org/10.1038/s41586-020-2012-7.Search in Google Scholar PubMed PubMed Central
6. Patel, VB, Zhong, JC, Grant, MB, Oudit, GY. Role of the ACE2/angiotensin 1–7 axis of the renin-angiotensin system in heart failure. Circ Res 2016;118:1313–26. https://doi.org/10.1161/circresaha.116.307708.Search in Google Scholar
7. Guo, YR, Cao, QD, Hong, ZS, Tan, YY, Chen, SD, Jin, HJ, et al. The origin, transmission and clinical therapies on coronavirus disease 2019 (COVID-19) outbreak - an update on the status. Mil Med Res 2020;7:11. https://doi.org/10.1186/s40779-020-00240-0.Search in Google Scholar PubMed PubMed Central
8. Shereen, MA, Khan, S, Kazmi, A, Bashir, N, Siddique, R. COVID-19 infection: origin, transmission, and characteristics of human coronaviruses. J Adv Res 2020;24:91–8. https://doi.org/10.1016/j.jare.2020.03.005.Search in Google Scholar PubMed PubMed Central
9. Mehra, MR, Siddiqi, HK. COVID-19 illness in native and immunosuppressed states: a clinical-therapeutic staging proposal. J Heart Lung Transplant 2020;39:405–7. https://doi.org/10.1016/j.healun.2020.03.012.Search in Google Scholar PubMed PubMed Central
10. Channappanavar, R, Fehr, AR, Vijay, R, Mack, M, Zhao, J, Meyerholz, DK, et al. Dysregulated type I interferon and inflammatory monocyte-macrophage responses cause lethal pneumonia in SARS-CoV-infected mice. Cell Host Microbe 2016;19:181–93. https://doi.org/10.1016/j.chom.2016.01.007.Search in Google Scholar PubMed PubMed Central
11. Pedersen, SF, Ho, Y-C. SARS-CoV-2: a storm is raging. J Clin Invest 2020;130:2202–5. https://doi.org/10.1172/jci137647.Search in Google Scholar
12. Lin, L, Lu, L, Cao, W, Li, T. Hypothesis for potential pathogenesis of SARS-CoV-2 infection-a review of immune changes in patients with viral pneumonia. Emerg Microbes Infect 2020;9:727–32. https://doi.org/10.1080/22221751.2020.1746199.Search in Google Scholar PubMed PubMed Central
13. Hu, W, Yen, YT, Singh, S, Kao, CL, Wu-Hsieh, BA. SARS-CoV regulates immune function-related gene expression in human monocytic cells. Viral Immunol 2012;25:277–88. https://doi.org/10.1089/vim.2011.0099.Search in Google Scholar PubMed PubMed Central
14. Channappanavar, R, Perlman, S. Pathogenic human coronavirus infections: causes and consequences of cytokine storm and immunopathology. Semin Immunopathol 2017;39:529–39. https://doi.org/10.1007/s00281-017-0629-x.Search in Google Scholar PubMed PubMed Central
15. McGonagle, D, Sharif, K, O’Regan, A, Bridgewood, C. The role of cytokines including interleukin-6 in COVID-19 induced pneumonia and macrophage activation syndrome-like disease. Autoimmun Rev 2020;19:102537. https://doi.org/10.1016/j.autrev.2020.102537.Search in Google Scholar PubMed PubMed Central
16. Wang, W, Ye, L, Ye, L, Li, B, Gao, B, Zeng, Y, et al. Up-regulation of IL-6 and TNF-α induced by SARS-coronavirus spike protein in murine macrophages via NF-κB pathway. Virus Res 2007;128:1–8. https://doi.org/10.1016/j.virusres.2007.02.007.Search in Google Scholar PubMed PubMed Central
17. García de Lorenzo y Mateos, A, López Martínez, J, Sánchez Castilla, M. Systemic inflammatory response: definitions, inflammatory markers and therapeutic possibilities. Med Intensiva. 2000;24:361–70. https://doi.org/10.1016/s0210-5691(00)79623-9.Search in Google Scholar
18. Chen, G, Wu, D, Guo, W, Cao, Y, Huang, D, Wang, H, et al. Clinical and immunologic features in severe and moderate coronavirus disease 2019. J Clin Invest 2020;130:2620–9. https://doi.org/10.1172/jci137244.Search in Google Scholar PubMed PubMed Central
19. Wen, W, Su, W, Tang, H, Le, W, Zhang, X, Zheng, Y, et al. Immune cell profiling of COVID-19 patients in the recovery stage by single-cell sequencing. Cell Discov 2020;6:31. https://doi.org/10.1038/s41421-020-00187-5.Search in Google Scholar PubMed PubMed Central
20. Thevarajan, I, Nguyen, T, Koutsakos, M, Druce, J, Caly, L, van de Sandt, C, et al. Breadth of concomitant immune responses prior to patient recovery: a case report of non-severe COVID-19. Nat Med 2020;26:453–5. https://doi.org/10.1038/s41591-020-0819-2.Search in Google Scholar PubMed PubMed Central
21. Wang, F, Nie, J, Wang, H, Zhao, Q, Xiong, Y, Deng, L, et al. Characteristics of peripheral lymphocyte subset alteration in COVID-19 pneumonia. J Infect Dis 2020;221:1762–9. https://doi.org/10.1093/infdis/jiaa150.Search in Google Scholar PubMed PubMed Central
22. Zheng, HY, Zhang, M, Yang, CX, Zhang, N, Wang, XC, Yang, XP, et al. Elevated exhaustion levels and reduced functional diversity of T cells in peripheral blood may predict severe progression in COVID-19 patients. Cell Mol Immunol 2020;17:541–3. https://doi.org/10.1038/s41423-020-0401-3.Search in Google Scholar PubMed PubMed Central
23. Janice Oh, H-L, Ken-En Gan, S, Betoletti, A, Tan, Y-J. Understanding the T cell immune response in SARS coronavirus infection. Emerg Microbes Infect 2012;1:e23. https://doi.org/10.1038/emi.2012.26.Search in Google Scholar PubMed PubMed Central
24. Zhao, J, Zhao, J, Mangalam, AK, Channappanavar, R, Fett, C, Meyerholz, DK, et al. Airway memory CD4+ T Cells mediate protective immunity against emerging respiratory coronaviruses. Immunity 2016;44:1379–91. https://doi.org/10.1016/j.immuni.2016.05.006.Search in Google Scholar PubMed PubMed Central
25. Li, CK, Wu, H, Yan, H, Ma, S, Wang, L, Zhang, M, et al. T cell responses to whole SARS coronavirus in humans. J Immunol 2008;181:5490–500. https://doi.org/10.4049/jimmunol.181.8.5490.Search in Google Scholar PubMed PubMed Central
26. Li, G, Chen, X, Xu, A. Profile of specific antibodies to the SARS-associated coronavirus. N Engl J Med 2003;349:508–9. https://doi.org/10.1056/nejm200307313490520.Search in Google Scholar
27. Li, X, Geng, M, Peng, Y, Meng, L, Lu, S. Molecular immune pathogenesis and diagnosis of COVID-19. J Pharm Anal 2020;10:102–8. https://doi.org/10.1016/j.jpha.2020.03.001.Search in Google Scholar PubMed PubMed Central
28. Guo, L, Ren, L, Yang, S, Xiao, M, Chang, D, Yang, F, et al. Profiling early humoral response to diagnose novel coronavirus disease (COVID-19). Clin Infect Dis 2020:1–28. https://doi.org/10.1093/cid/ciaa310.Search in Google Scholar PubMed PubMed Central
29. Long, QX, Liu, BZ, Deng, HJ, Wu, GC, Deng, K, Chen, YK, et al. Antibody responses to SARS-CoV-2 in patients with COVID-19. Nat Med 2020 Apr 29. https://doi.org/10.1038/s41591-020-0897-1 [Epub antes de impresión].Search in Google Scholar PubMed
30. Zhao, J, Yuan, Q, Wang, H, Liu, W, Liao, X, Su, Y, et al. Antibody responses to SARS-CoV-2 in patients of novel coronavirus disease 2019. Clin Infect Dis 2020 Mar 28. https://doi.org/10.1093/cid/ciaa344 [Epub antes de impresión].Search in Google Scholar PubMed PubMed Central
31. Zhang, B, Zhou, X, Zhu, C, Feng, F, Qiu, Y, Feng, J, et al. Immune phenotyping based on neutrophil-to-lymphocyte ratio and IgG predicts disease severity and outcome for patients with COVID-19. medRxiv 2020 Mar 16. https://doi.org/10.2020/.03.12.20035048.[Preprint not peer reviewed].10.1101/2020.03.12.20035048Search in Google Scholar
32. Bosmann, M, Ward, PA. Role of C3, C5 and anaphylatoxin receptors in acute lung injury and in sepsis. Adv Exp Med Biol 2012;946:147–59. https://doi.org/10.1007/978-1-4614-0106-3_9.Search in Google Scholar PubMed PubMed Central
33. Xiang, F, Wang, X, He, X, Peng, Z, Yang, B, Zhang, J, et al. Antibody detection and dynamic characteristics in patients with COVID-19. Clin Infect Dis 2020 Apr 19. https://doi.org/10.1093/cid/ciaa461 [Epub antes de impresión].Search in Google Scholar PubMed PubMed Central
34. Iwasaki, A, Yang, Y. The potential danger of suboptimal antibody responses in COVID-19. Nat Rev Immunol 2020;20:339–41. https://doi.org/10.1038/s41577-020-0321-6.Search in Google Scholar PubMed PubMed Central
35. Loeffelholz, MJ, Tang, YW. Laboratory diagnosis of emerging human coronavirus infections – the state of the art. Emerg Microbes Infect 2020;9:747–56. https://doi.org/10.1080/22221751.2020.1745095.Search in Google Scholar PubMed PubMed Central
36. Li, D, Wang, D, Dong, J, Wang, N, Huang, H, Xu, H, et al. False-negative results of real-time reverse-transcriptase polymerase chain reaction for severe acute respiratory syndrome coronavirus 2: role of deep-learning-based ct diagnosis and insights from two cases. Korean J Radiol 2020;21:505–8. https://doi.org/10.3348/kjr.2020.0146.Search in Google Scholar PubMed PubMed Central
37. Chen, Z, Li, Y, Wu, B, Hou, Y, Bao, J, Deng, X. A patient with COVID-19 presenting a false-negative reverse transcriptase polymerase chain reaction result. Korean J Radiol 2020;21:623–4. https://doi.org/10.3348/kjr.2020.0195.Search in Google Scholar PubMed PubMed Central
38. Patel, R, Babady, E, Theel, ES, Storch, GA, Pinsky, BA, George, KS, et al. Report from the American Society for Microbiology COVID-19: value of diagnostic testing for SARS–CoV-2/COVID-19. Am Soc Microbiol 2020;11:1–5. https://doi.org/10.1128/mbio.00722-20.Search in Google Scholar PubMed PubMed Central
39. Xie, X, Zhong, Z, Zhao, W, Zheng, C, Wang, F, Liu, J. Chest CT for typical 2019-nCoV pneumonia: relationship to negative RT-PCR testing. Radiology 2020 Feb 12. https://doi.org/10.1148/radiol.2020200343 [Epub antes de impresión].Search in Google Scholar PubMed PubMed Central
40. Diao, B, Wen, K, Chen, J, Liu, Y, Yuan, Z, Han, C, et al. Diagnosis of acute respiratory syndrome coronavirus 2 infection by detection of nucleocapsid protein. medRxiv. 2020 Mar 13. https://doi.org/10.1101/2020.03.07.20032524 [Preprint not peer reviewed].Search in Google Scholar
41. Li, Z, Yi, Y, Luo, X, Xiong, N, Liu, Y, Li, S, et al. Development and clinical application of a rapid IgM-IgG combined antibody test for SARS-CoV-2 infection diagnosis. J Med Virol 2020 Feb 24. https://doi.org/10.1002/jmv.25727 [Epub antes de impresión].Search in Google Scholar PubMed PubMed Central
42. Zhang, W, Du, RH, Li, B, Zheng, XS, Yang, XL, Hu, B, et al. Molecular and serological investigation of 2019-nCoV infected patients: implication of multiple shedding routes. Emerg Microbes Infect 2020;9:386–9. https://doi.org/10.1080/22221751.2020.1729071.Search in Google Scholar PubMed PubMed Central
43. Portal de Salud de la Junta de Castilla y León. Guía breve para la utilización de test diagnósticos en Infección por COVID 19 [Internet]. Disponible en: https://www.saludcastillayleon.es/es/covid-19/informacion-profesionales/ultimas-novedades/guia-breve-utilizacion-test-diagnosticos-infeccion-covid-19 [Fecha de consulta 10 Abr 2020].Search in Google Scholar
44. Sociedad Española de Inmunología. Anticuerpos anti SARS-CoV-2 Propuesta de implementación como prueba diagnóstica, pronóstica y de desarrollo de inmunidad protectora [Internet]. Barcelona: SEI - Sociedad Española de Inmunología; 2020 Abr 02 [cited 2020 Jun 13]. Disponible en: https://inmunologia.org/covid19/fitxa_noticia.php?UpRu5Am=HLID.Search in Google Scholar
45. Bao, L, Deng, W, Gao, H, Xiao, C, Liu, J, Xue, J, et al. Reinfection could not occur in SARS-CoV-2 infected rhesus macaques. bioRxiv 2020 Mar 14. https://doi.org/10.1101/2020.03.13.990226 [Preprint not peer reviewed].Search in Google Scholar
46. FIND [Internet]. 2020 [cited: 2020 May 5]. Disponible en: https://www.finddx.org/.Search in Google Scholar
47. Lassaunière, R, Frische, A, Harboe, ZB, Nielsen, ACY, Fomsgaard, A, Karen, A, et al. Evaluation of nine commercial SARS-CoV-2 immunoassays. MedRxiv 2020 Apr 10. https://doi.org/10.1101/2020.04.09.20056325 [Preprint not peer reviewed].Search in Google Scholar
48. Whitman, JD, Hiatt, J, Mowery, CT, Shy, BR, Yu, R, Yamamoto, TN, et al. Test performance evaluation of SARS-CoV-2 serological assays. MedRxiv 2020 Apr 29. https://doi.org/10.1101/2020.04.25.20074856 [Preprint not peer reviewed].Search in Google Scholar PubMed PubMed Central
49. Lin, D, Liu, L, Mingxia, Z, Hu, Y, Yang, Q, Guo, J, et al. Evaluations of serological test in the diagnosis of 2019 novel coronavirus (SARS-CoV-2) infections during the COVID-19 outbreak. MedRxiv 2020 Mar 30. https://doi.org/10.1101/2020.03.27.20045153 [Preprint not peer reviewed].Search in Google Scholar
50. Favalli, EG, Ingegnoli, F, De Lucia, O, Cincinelli, G, Cimaz, R, Caporali, R. COVID-19 infection and rheumatoid arthritis: faraway, so close!. Autoimmun Rev 2020;19:102523. https://doi.org/10.1016/j.autrev.2020.102523.Search in Google Scholar PubMed PubMed Central
51. Yao, TT, Qian, JD, Zhu, WY, Wang, Y, Wang, GQ. A systematic review of lopinavir therapy for SARS coronavirus and MERS coronavirus—a possible reference for coronavirus disease-19 treatment option. J Med Virol 2020;92:556–63. https://doi.org/10.1002/jmv.25729.Search in Google Scholar PubMed PubMed Central
52. Davies, M, Osborne, V, Lane, S, Roy, D, Dhanda, S, Evans, A, et al. Remdesivir in treatment of COVID-19: a systematic benefit-risk assessment. Drug Saf 2020 May 28. https://doi.org/10.1007/s40264-020-00952-1 [Epub antes de impresión].Search in Google Scholar PubMed PubMed Central
53. Devaux, CA, Rolain, JM, Colson, P, Raoult, D. New insights on the antiviral effects of chloroquine against coronavirus: what to expect for COVID-19?. Int J Antimicrob Agents 2020;55:105938. https://doi.org/10.1016/j.ijantimicag.2020.105938.Search in Google Scholar PubMed PubMed Central
54. Generalitat de Catalunya. Recull de l’evidència del tractament farmacològic de la infecció per SARS-CoV-2 [Internet]. Disponible en: https://scientiasalut.gencat.cat/handle/11351/4881 [Fecha de consulta 26 May 2020].Search in Google Scholar
55. Xu, F, Wen, Z, Shi, X, Fan, J. Inflammasome in the pathogenesis of pulmonary diseases. Experientia Suppl 2018;108:111–51. https://doi.org/10.1007/978-3-319-89390-7_6.Search in Google Scholar PubMed PubMed Central
56. Zhang, C, Wu, Z, Li, JW, Zhao, H, Wang, GQ. Cytokine release syndrome in severe COVID-19: interleukin-6 receptor antagonist tocilizumab may be the key to reduce mortality. Int J Antimicrob Agents 2020;55:105954. https://doi.org/10.1016/j.ijantimicag.2020.105954.Search in Google Scholar PubMed PubMed Central
57. Russell, CD, Millar, JE, Baillie, JK. Clinical evidence does not support corticosteroid treatment for 2019-nCoV lung injury. Lancet 2020;395:473–5. https://doi.org/10.1016/s0140-6736(20)30317-2.Search in Google Scholar
58. Ministerio de Sanidad. Manejo clínico del COVID-19: atencion hospitalaria. Médico [Internet]. España: Ministerio de Sanidad; 2020 May [cited 2020 May 26]:23p. Disponible en: https://www.mscbs.gob.es/en/profesionales/saludPublica/ccayes/alertasActual/nCov-China/documentos/Protocolo_manejo_clinico_ah_COVID-19.pdf.Search in Google Scholar
59. Stebbing, J, Phelan, A, Griffin, I, Tucker, C, Oechsle, O, Smith, D, et al. COVID-19: combining antiviral and anti-inflammatory treatments. Lancet Infect Dis 2020;20:400–2. https://doi.org/10.1016/s1473-3099(20)30132-8.Search in Google Scholar PubMed PubMed Central
60. Ng, O, Chia, A, Tan, A, Jadi, R, Leong, H, Bertoletti, A, et al. Memory T cell responses targeting the SARS coronavirus persist up to 11 years postinfection. Vaccine 2016;34:2008–14. https://doi.org/10.1016/j.vaccine.2016.02.063.Search in Google Scholar PubMed PubMed Central
61. Monteagudo, L, Boothby, A, Gertner, E. Continuous intravenous anakinra infusion to calm the cytokine storm in macrophage activation syndrome. ACR Open Rheumatol 2020;2:276–82. https://doi.org/10.1002/acr2.11135.Search in Google Scholar PubMed PubMed Central
62. Chen, L, Xiong, J, Bao, L, Shi, Y. Convalescent plasma as a potential therapy for COVID-19. Lancet Infect Dis 2020;20:398–400. https://doi.org/10.1016/s1473-3099(20)30141-9.Search in Google Scholar PubMed PubMed Central
63. Shanmugaraj, B, Siriwattananon, K, Wangkanont, K, Phoolcharoen, W. Perspectives on monoclonal antibody therapy as potential therapeutic intervention for coronavirus disease-19 (COVID-19). Asian Pac J allergy Immunol 2020;38:10–8. https://doi.org/10.12932/AP-200220-0773.Search in Google Scholar PubMed
64. Agencia Española de Medicamentos y Productos Sanitarios. Tratamientos disponibles para el manejo de la infección respiratoria por SARS-CoV-2-Agencia Española de Medicamentos y Productos Sanitarios [Internet]. Disponible en: https://www.aemps.gob.es/laaemps/ultima-informacion-de-la-aemps-acerca-delcovid%E2%80%9119/tratamientos-disponibles-para-el-manejo-de-la-infeccionrespiratoria-por-sars-cov-2/?lang=ca [Fecha de consulta 26 May 2020].Search in Google Scholar
65. WHO. DRAFT landscape of COVID-19 candidate vaccines – 20 April 2020 [Internet]. Switzerland: WHO; 2020 April [cited 2020 May 26]: 7p. Available from: https://www.who.int/blueprint/priority-diseases/key-action/novelcoronavirus-landscape-ncov.pdf?ua=.Search in Google Scholar
66. Dhama, K, Sharun, K, Tiwari, R, Dadar, M, Malik, YS, Singh, KP, et al. COVID-19, an emerging coronavirus infection: advances and prospects in designing and developing vaccines, immunotherapeutics, and therapeutics. Hum Vaccin Immunother 2020 Mar 18. https://doi.org/10.1080/21645515.2020.1735227 [Epub antes de impresión].Search in Google Scholar PubMed PubMed Central
67. Letko, M, Marzi, A, Munster, V. Functional assessment of cell entry and receptor usage for SARS-CoV-2 and other lineage B betacoronaviruses. Nat Microbiol 2020;5:562–9. https://doi.org/10.1038/s41564-020-0688-y.Search in Google Scholar PubMed PubMed Central
68. Thanh Le, T, Andreadakis, Z, Kumar, A, Gómez, R, Tollefsen, S, Saville, M, et al. The COVID-19 vaccine development landscape. Nat Rev Drug Discov 2020;19:305–6. https://doi.org/10.1038/d41573-020-00073-5.Search in Google Scholar PubMed
69. Callaway, E. Vaccines the race for coronavirus vaccines. Nature 2020;580:576–7. https://doi.org/10.1038/d41586-020-01221-y.Search in Google Scholar PubMed
70. Amanat, F, Krammer, F. SARS-CoV-2 vaccines: status report. Immunity 2020;52:583–9. https://doi.org/10.1016/j.immuni.2020.03.007.Search in Google Scholar PubMed PubMed Central
© 2020 Cristina A. López Rodríguez et al., published by De Gruyter, Berlin/Boston
This work is licensed under the Creative Commons Attribution 4.0 International License.
Articles in the same Issue
- Editorial
- Brief update on coronavirus disease 2019 (COVID-19) diagnostics
- Breve actualización sobre el diagnóstico de la enfermedad por coronavirus 2019 (COVID-19)
- Challenges of COVID-19
- Retos de la COVID-19
- Review / Artículo de Revisión
- SARS-CoV-2: a new threat
- SARS-CoV-2: una nueva amenaza
- VALIDA project: Validation of allergy in vitro diagnostics assays (Tools and recommendations for the assessment of in vitro tests in the diagnosis of allergy)
- Proyecto VALIDA: Validation of ALlergy In vitro Diagnostics Assays (Herramientas y recomendaciones para la valoración de las pruebas in vitro en el diagnóstico de la alergia)
- Original Article / Artículo Original
- Biomaker evaluation for major adverse cardiovascular event development in patients undergoing cardiac Surgery
- Evaluación del papel de diversos biomarcadores en el desarrollo de eventos cardiovasculares adversos mayores en pacientes sometidos a cirugía cardíaca
- CYFRA 21-1 in patients with suspected cancer: evaluation of an optimal cutoff to assess the diagnostic efficacy and prognostic value
- El CYFRA 21-1 en pacientes con sospecha de cáncer: evaluación de un punto de corte óptimo para evaluar la eficacia diagnóstica y el valor pronóstico
- Lactose tolerance test as an alternative to hydrogen breath test in the study of lactose malabsorption
- Estudio del test de tolerancia a lactosa como alternativa a test de hidrógeno espirado en el estudio de la malabsorción de lactosa
- Precision and trueness verification study of an Atellica system
- Estudio de verificación de precisión y veracidad en un sistema Atellica
- Case Report / Caso Clínico
- A variant of the gene HARS detected in the clinical exome: etiology of a peripheral neuropathy undiagnosed for 20 years
- Variante en gen HARS detectada en exoma clínico: etiología de neuropatía periférica tras más de 20 años sin diagnóstico
- Letter to the Editor / Carta al Editor
- Stability of lactate dehydrogenase in plasma at different temperatures: post-analytical storage
- Estudio de la estabilidad de la actividad lactato deshidrogenasa en plasma a distintas temperaturas: conservación post-analítica
Articles in the same Issue
- Editorial
- Brief update on coronavirus disease 2019 (COVID-19) diagnostics
- Breve actualización sobre el diagnóstico de la enfermedad por coronavirus 2019 (COVID-19)
- Challenges of COVID-19
- Retos de la COVID-19
- Review / Artículo de Revisión
- SARS-CoV-2: a new threat
- SARS-CoV-2: una nueva amenaza
- VALIDA project: Validation of allergy in vitro diagnostics assays (Tools and recommendations for the assessment of in vitro tests in the diagnosis of allergy)
- Proyecto VALIDA: Validation of ALlergy In vitro Diagnostics Assays (Herramientas y recomendaciones para la valoración de las pruebas in vitro en el diagnóstico de la alergia)
- Original Article / Artículo Original
- Biomaker evaluation for major adverse cardiovascular event development in patients undergoing cardiac Surgery
- Evaluación del papel de diversos biomarcadores en el desarrollo de eventos cardiovasculares adversos mayores en pacientes sometidos a cirugía cardíaca
- CYFRA 21-1 in patients with suspected cancer: evaluation of an optimal cutoff to assess the diagnostic efficacy and prognostic value
- El CYFRA 21-1 en pacientes con sospecha de cáncer: evaluación de un punto de corte óptimo para evaluar la eficacia diagnóstica y el valor pronóstico
- Lactose tolerance test as an alternative to hydrogen breath test in the study of lactose malabsorption
- Estudio del test de tolerancia a lactosa como alternativa a test de hidrógeno espirado en el estudio de la malabsorción de lactosa
- Precision and trueness verification study of an Atellica system
- Estudio de verificación de precisión y veracidad en un sistema Atellica
- Case Report / Caso Clínico
- A variant of the gene HARS detected in the clinical exome: etiology of a peripheral neuropathy undiagnosed for 20 years
- Variante en gen HARS detectada en exoma clínico: etiología de neuropatía periférica tras más de 20 años sin diagnóstico
- Letter to the Editor / Carta al Editor
- Stability of lactate dehydrogenase in plasma at different temperatures: post-analytical storage
- Estudio de la estabilidad de la actividad lactato deshidrogenasa en plasma a distintas temperaturas: conservación post-analítica

